Codificando el futuro: Bacterias como memorias vivientes
Karen Lizeth Sánchez Rodríguez

Figura 1. Representación conceptual de bacterias como unidades de memoria. Imagen generada por inteligencia artificial con Meta AI. Creación original del Dr. Pavel Andreí Montero Domínguez, 2025.
En la década de 1980 inició lo que conocemos como "la era digital", con el desarrollo y comercialización de los primeros microprocesadores, que fueron importantes en el desarrollo de las tecnologías de la información. Sin embargo, este progreso nos enfrenta al desafío de la gestión y almacenamiento de dicha información. Esto último fue evidenciado en la obtención de la primera foto de un agujero negro en el 2019 [1]. Para esta hazaña, se requirió un total de 4 mil terabytes, lo que equivale a 31,250 iPhones 16 Pro de 128 Gb de almacenamiento para la captura, el procesamiento y la producción de esta icónica imagen (figura 2).

Figura 2. Representación visual de 31,250 iPhones comparados con la escala de un humano promedio. Imagen generada por inteligencia artificial mediante Meta AI. Creación original de la autora, 2025.
Desde su creación, los discos duros han sido dispositivos utilizados para el almacenamiento de datos, no obstante, la velocidad de generación de información por parte del ser humano ha dejado en entredicho la viabilidad de su uso futuro. El principal problema es la capacidad y cantidad de discos utilizados, incrementado con ello el costo total de la configuración y resultando prohibitivo para algunos usuarios. Por otro lado, el mantenimiento y el consumo energético para su uso y refrigeración son elevados, y algunos discos pueden alcanzar hasta 80 °C en condiciones de alta carga de trabajo. Falta de ventilación adecuada, entornos sin refrigeración y uso prolongado sin descanso, son factores que implican gastos considerables para algunas empresas.
Esto último ha llevado a compañías como Microsoft a explorar soluciones innovadoras para enfrentar los desafíos del consumo de energía, como lo es el proyecto Natick [2]. Cuando se envía información a la 'nube', ésta termina alojada en estructuras físicas conocidas como servidores. En un esfuerzo por mejorar la estabilidad y la eficiencia energética de éstos, Microsoft ha implementado la idea de sumergir estos servidores en el océano (figura 3).

Figura 3. Servidor de almacenamiento de datos de la empresa Microsoft siendo sumergido en el océano. Imagen tomada de BBC News Mundo. [¿Por qué Microsoft instaló un centro de datos en el fondo del océano? - BBC News Mundo](https://www.bbc.com/mundo/noticias/2016/02/160202microsoftcentrodatosdebajoaguamarsubaminoall)
Aunque esta propuesta parece prometedora, aún se encuentra en fase de prueba. Durante sus primeras pruebas, aproximadamente una octava parte de los servidores experimentaron fallos debido a variaciones en la temperatura, humedad y oxidación. Por otro lado, se sabe que las altas concentraciones de metales y metaloides tienen efectos tóxicos en los organismos acuáticos [3]. Asimismo, debemos tomar en cuenta el impacto ambiental causado por la obtención de la materia prima para construir estas unidades de almacenamiento, como es el caso de los metales en la industria minera. Estos conllevan graves consecuencias ambientales, como la remoción de grandes cantidades de suelo y roca, lo que resulta en la degradación del suelo, la pérdida de biodiversidad y la contaminación del agua. Por otra parte, un artículo del periódico El Economista [4] reporta que la producción de una tonelada de aluminio requiere de 14 megavatios-hora de energía, lo cual es equivalente a aproximadamente 120 veces el consumo per cápita mensual en Cuernavaca [5]. Esto se traduce en un impacto económico y ambiental considerable a tomar en cuenta.
Considerando todo lo anterior, ¿qué pensarías si te dijera que existe una alternativa biológica para resolver esta problemática? Una idea un poco loca, ¿no crees? Un estudio publicado en la revista Nature por investigadores de la Universidad de Harvard (USA), reveló que se puede almacenar información visual (imágenes y un cortometraje) en un organismo vivo [6]. ¿Cómo lo consiguieron? En este trabajo lograron codificar una representación bidimensional de una mano, que frecuentemente se almacena en código binario, en el código genético de bacterias, específicamente en Escherichia coli (E. coli).
¿Te imaginas si te dijera que no sólo se logró almacenar una imagen de una mano? Mucho antes, en 1878, se capturaron las distintas etapas del galope de un caballo, creando uno de los primeros GIFs (“imágenes en movimiento”) conocidos [7]. Este pequeñovideo, compuesto por no más de 16 fotogramas, se convirtió en un hito en la historia de la fotografía y el cine. Melancolía o no, este fue el GIF seleccionado para codificarlo en el código genético de E. coli, logrando con ello pasar nuevamente a la historia al ser no sólo el primer cortometraje creado, sino el primero reconstruido a partir del ADN de una bacteria (figura 4).
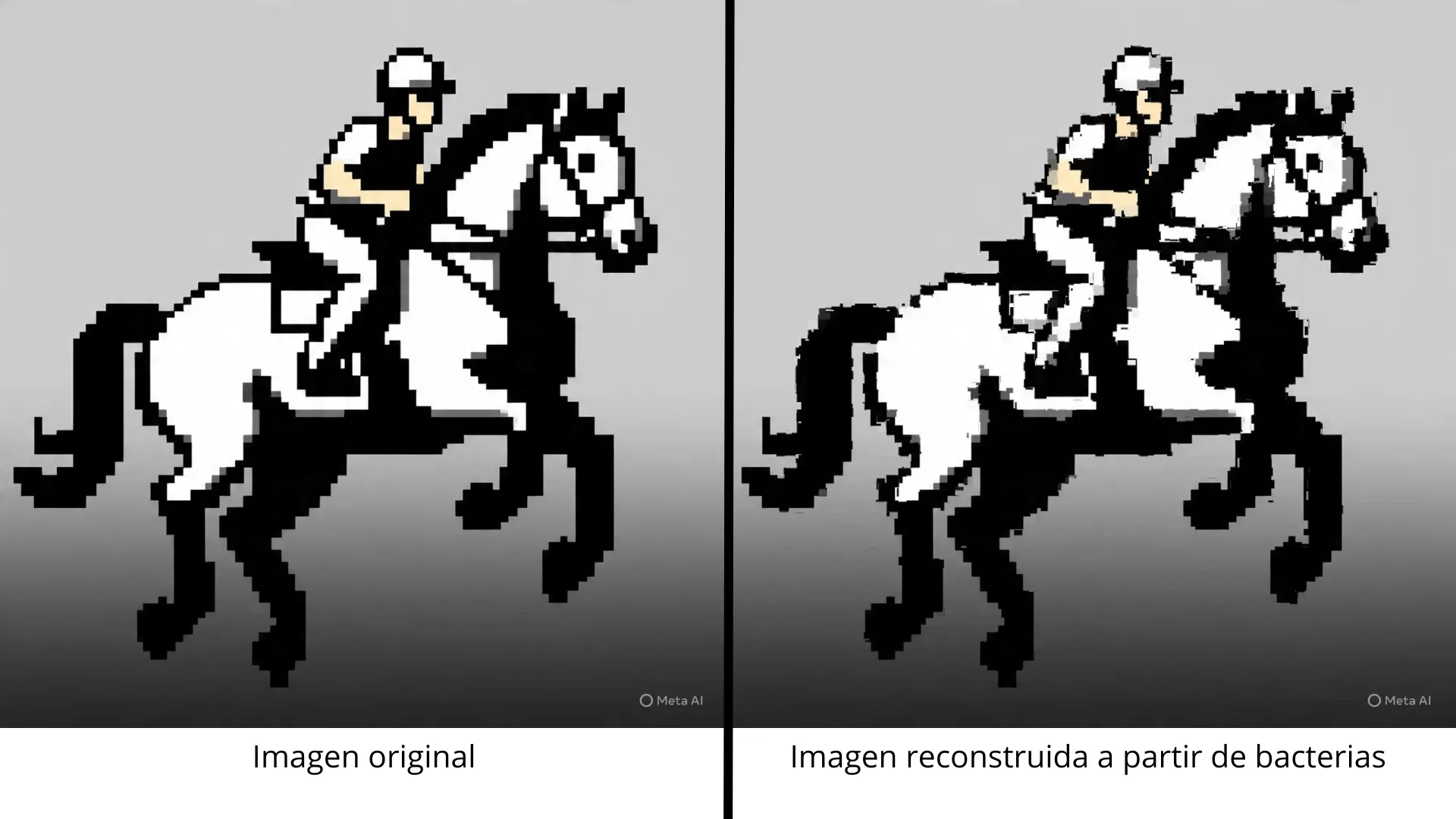
Figura 4. Comparación entre la imagen original y reconstrucción bacteriana. Imágenes generadas por inteligencia artificial mediante Meta IA como reinterpretación de la figura publicada en Shipman et al. [6]. Creación original del Dr. Pavel Andreí Montero Domínguez, 2025.
Todo este hito fue logrado gracias al famoso sistema CRISPR-Cas (ver Biotecnología en Movimiento, número 35-5: https://biotecmov.ibt.unam.mx/numeros/35/5.html), el cual permite modificar el genoma de cualquier célula (bacteria), al eliminar o añadir el gen deseado. Mientras las computadoras utilizan el código binario (0 y 1), los organismos vivos almacenan su información en cadenas de ADN, siendo las bases de éstas, las moléculas Adenina (A), Timina (T), Guanina (G) y Citosina (C). Pudiendo entender esto, los investigadores únicamente convirtieron la información del código binario del contenido visual, al código de la vida (ADN) para su posterior inserción en el genoma del organismo deseado (Figura 5).
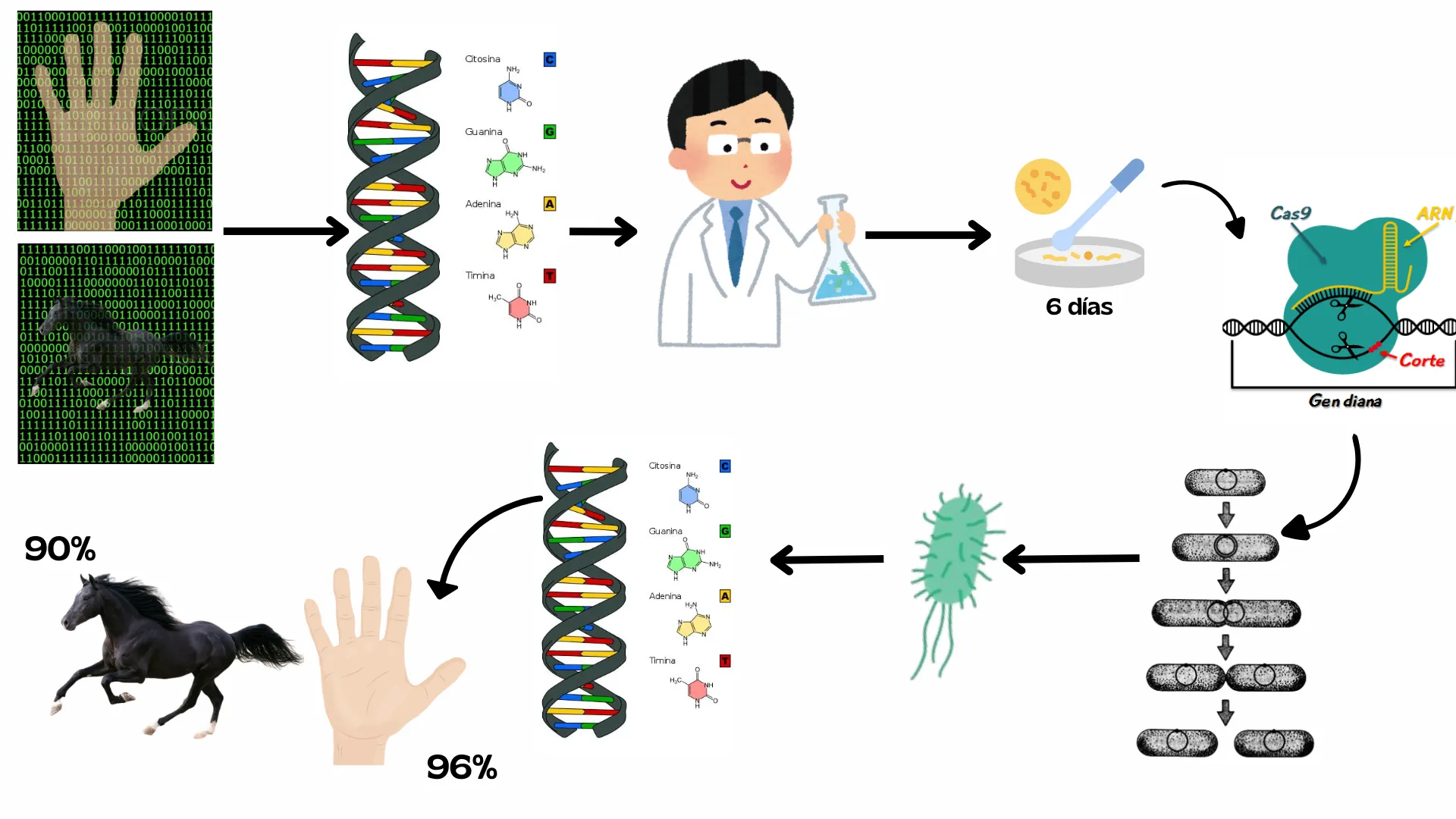
Figura 5. Esquema de cómo se llevó a cabo el experimento de Shipman y colaboradores [6].
Una vez hecho esto, se cultivaron las bacterias durante 6 días, lo que en E. coli equivale a 48 generaciones. Imagina que tu tatara tatara tatara tatara abuelo te diera una carta con una información clave y tú, en la actualidad, la pudieras leer tal y como la escribió tu ancestro. Exactamente eso fue lo que hicieron para lograr obtener la información con un 90 % de su resolución original, para el caso del GIF, y en el caso de la mano, con un 96% de su resolución y fidelidad (Figura 6) [6].
Si bien es una tecnología en desarrollo y aún se requiere evaluar su viabilidad económica y energética para su uso, la posibilidad del uso de organismos biológicos como unidades de almacenamiento de la información, parece presentar grandes ventajas. Siendo sus principales ventajas su capacidad de auto-replicarse y su naturaleza biodegradable. Esta idea se ha propuesto como una alternativa interesante para su posible uso en el futuro, pudiendo ser esta tecnología de almacenamiento, el próximo avance revolucionario en la informática, como lo fue la invención de la computadora, hace 88 años.

Figura 6. Artículo del cual se tomó la información para este ensayo. Tomado de [7]. Disponible en: https://doi.org/10.1038/nature23017
Referencias
- Juárez, C. (2029) “La primera imagen de un hoyo negro real”. https://ciencia.unam.mx/leer/854/la-primera-imagen-de-un-hoyo-negro-real.
- Microsoft Prensa, “Proyecto Natick, el futuro de los centros de datos bajo el mar es fiable, práctico y sostenible” – Centro de noticias. (2020, Septiembre 15). Microsoft News. Recuperado el 15 de abril de 2024, de: https://news.microsoft.com/es-es/2020/09/15/proyecto-natick-el-futuro-de-los-centros-de-datos-bajo-el-mar-es-fiable-practico-y-sostenible/
- Santillán, M. L. (2022, mayo 19). “¿Cómo afecta la contaminación a los ecosistemas costeros”? Ciencia UNAM. Recuperado el 15 de Abril de 2024, de: https://ciencia.unam.mx/leer/1263/-como-afecta-la-contaminacion-a-los-ecosistemas-costeros-
- Blanco, V., & Martínez, M. (2021, octubre 12). “Ya cuesta igual la tonelada de aluminio que la electricidad para producirla”. elEconomista.es. Recuperado el 15 de Abril de 2024, de: https://www.eleconomista.es/empresas-finanzas/noticias/11427917/10/21/Ya-cuesta-igual-la-tonelada-de-aluminio-que-la-electricidad-para-producirla-.html
- Salgado Arcos, J. A. (n.d.). Reporte interno del Centro de Estudios Demográficos y Ambientales. “Análisis de la distribución del consumo doméstico de energía eléctrica en la Zona Metropolitana de Cuernavaca, Morelos”, https://cedua.colmex.mx/archivos/458/a42-jesus-salgado-.pdf
- Shipman, S. L., Nivala, J., Macklis, J. D., & Church, G. M. (2017, Julio 12). CRISPR–Cas encoding of a digital movie into the genomes of a population of living bacteria. Nature, 547, 14. NOTA: Debo admitir que sí me fue complicado entender esta referencia, pero recibí apoyo por parte del profesor de microbiología, el Dr. Pavel Andreí; de mi profesor de Biotecnología, Alid Guadarrama y de la maestra Karina Rangel, los cuales, además de ser profesores de la PREFECO A.Q.R., forman parte del Instituto de Biotecnología de la UNAM. Además, con las materias cursadas en la preparatoria (Biología 1 y 2, Microbiología, Operaciones Unitarias y Biotecnología) me permitieron comprender algunos términos, facilitando su comprensión. DOI: 10.1038/nature23017.
- Shah, H. (13 de diciembre de 2018), “Cómo un fotógrafo del siglo XIX creó el primer GIF de un caballo al galope”, Smithsonian Magazine, https://www.smithsonianmag.com/smithsonian-institution/how-19th-century-photographer-first-gif-galloping-horse-180970990/
Comparte este artículo en redes sociales

Acerca de los autores
Karen Lizeth, al momento de escribir este texto, tenía 18 años y era alumna del 6° semestre, en la Preparatoria Federal por Cooperación “Andrés Quintana Roo”, de Chamilpa, Morelos, en la capacitación de Laboratorista Químico. Es de Cuernavaca, Morelos y ganó el 1er y 3er lugar en la materia de Biología de “Inter-PREFECOS” a nivel estatal y nacional. Actualmente cursa la Lic. en Ciencias del área terminal Bioquímica y Biología Molecular en el Centro de Investigación en Dinámica Celular, IICBA, de la Universidad Autónoma del Estado de Morelos (UAEM).
Contacto: karen.sanchez5097828@gmail.com

