Descifrando a un enemigo diminuto
M. en C. Isela Serrano Fujarte, Biól. Jimena Berenice García Domínguez y Dr. José Luis Puente García
Las bacterias son organismos vivos diminutos, invisibles a nuestros ojos, capaces de habitar en diversos entornos, como el agua, el suelo y nuestros propios cuerpos. Algunos de estos microorganismos son benéficos, mientras que otros pueden provocar enfermedades y se les considera patógenos. Tal es el caso de Salmonella enterica subsp. enterica sv. Typhimurium (S. Typhimurium), un nombre muy imponente y complejo para un organismo de tamaño minúsculo, pero en línea con sus sofisticadas habilidades (Figura 1).
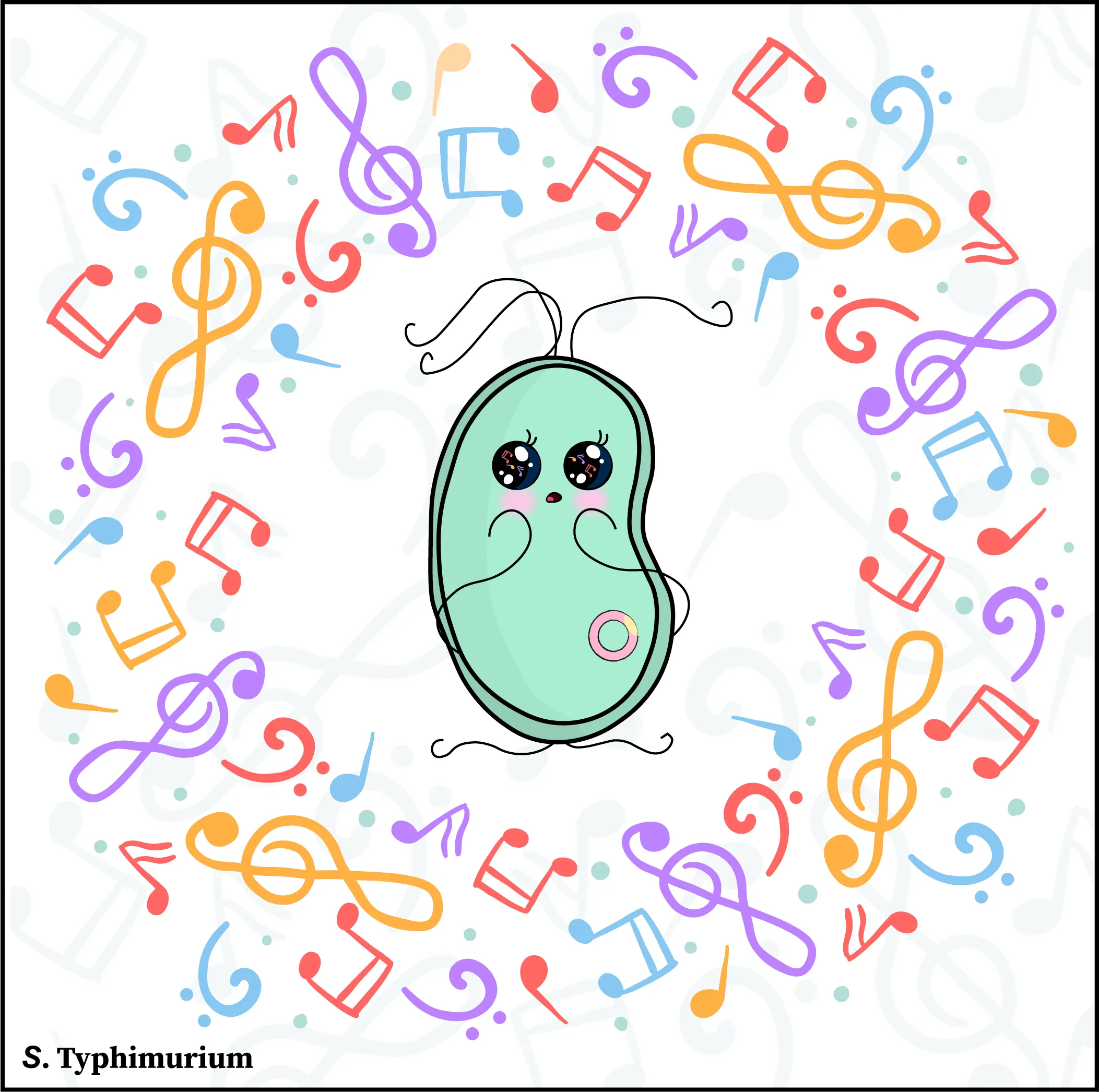
Figura 1. Las bacterias, tanto las patógenas como las benéficas, nos han guiado a través de los recovecos del conocimiento. Han servido como modelos experimentales, desvelando los secretos de la genética y la biología molecular, enseñándonos sobre adaptación y evolución. Dejándonos lecciones invaluables sobre la vida, la salud y la complejidad del cosmos microscópico.
Esta bacteria puede sobrevivir en el ambiente mediante la formación de biopelículas, una especie de fortalezas protectoras que le permiten resistir condiciones adversas, tales como la desecación, la luz ultravioleta, los desinfectantes y los antibióticos. No obstante, este mecanismo también le faculta para subsistir y, posteriormente, ser ingerida y causar enfermedades gastrointestinales e invasivas tanto en humanos como en animales.
Para lograr tal cometido, cuando estos minúsculos microorganismos se adentran en el cuerpo humano durante la infección, en un acto de subversión biológica, despliegan un repertorio de tácticas moleculares sumamente astutas para apoderarse de las funciones celulares y redirigir los procesos normales del organismo en su propio beneficio.
Primero se introduce en las células usando una jeringa molecular que inyecta a la célula proteínas de la bacteria que la obligan a abrir una puerta de entrada. Una vez en el interior, se aloja en un compartimento especial que le brinda protección contra las defensas celulares, lo que le permite sobrevivir y replicarse. Además de estas características, esta bacteria destaca por su habilidad para escapar y ocultarse, lo que le permite alcanzar el torrente sanguíneo, diseminarse por el organismo y provocar enfermedades invasivas potencialmente letales. Estas enfermedades pueden ser especialmente graves en infantes o personas con sistemas inmunológicos debilitados (Figura 2).
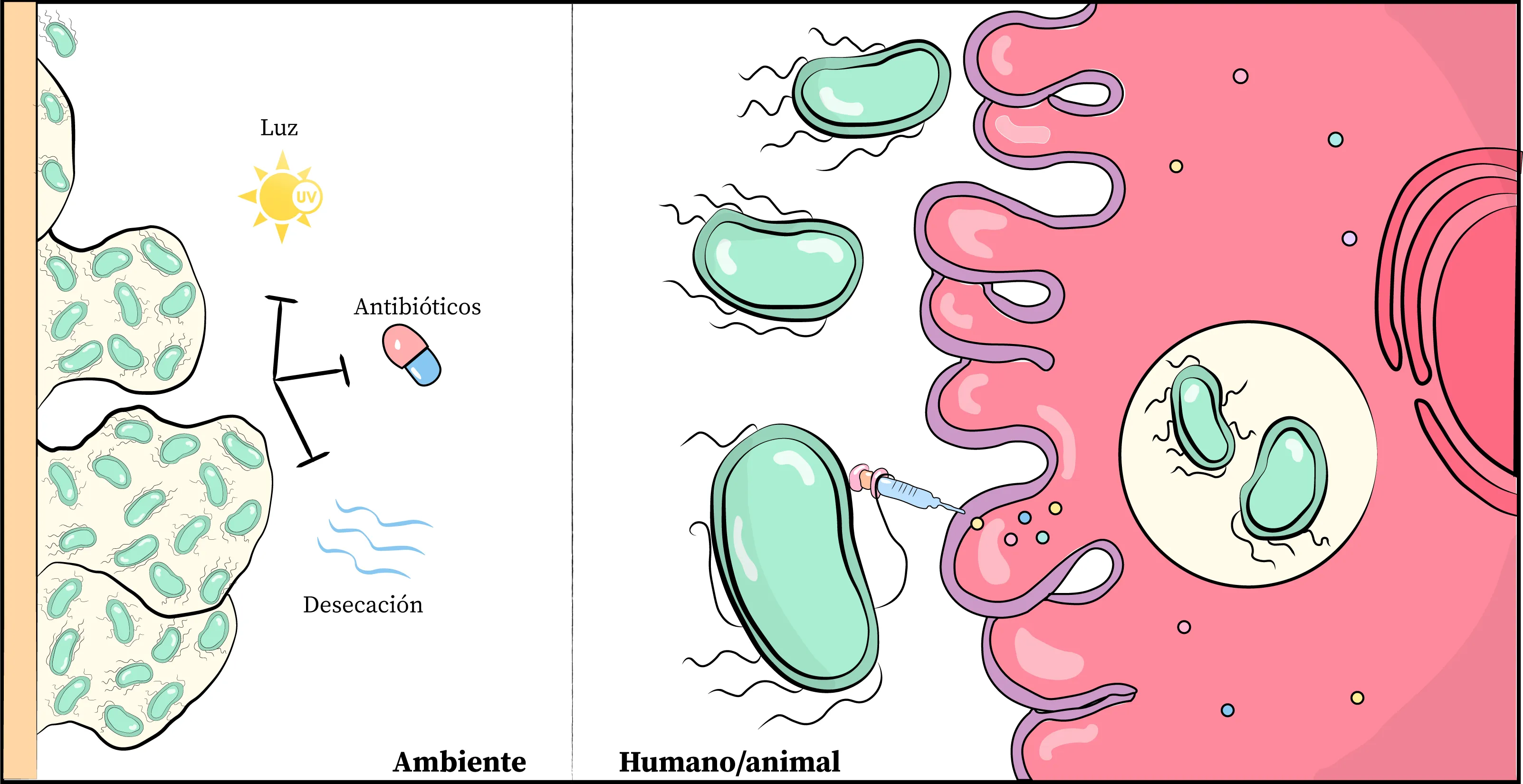
Figura 2. S. Typhimurium sobrevive en el ambiente formando biopelículas que le brindan protección ante las inclemencias del entorno. Luego puede ser ingerida, desencadenando infecciones en seres humanos y animales, para ello despliega una “jeringa” molecular con la que inyecta proteínas a la célula, las cuales le permiten tomar el control de la maquinaria celular, alterando el curso de las funciones celulares en su propio beneficio.
El origen
S. Typhimurium parece el arquetipo de un villano digno de la gran pantalla, pero no siempre fue así. En la época dorada de la bacteriología, una rama científica dedicada al estudio de las bacterias, un joven investigador alemán llamado Friedrich Loeffler se encontró con el agente causal de la fiebre tifoidea en ratones. No imaginaría que su trabajo con este microorganismo lo llevaría a emprender una travesía científica por los campos de Tesalia en Grecia.
Loeffler descubrió que S. Typhimurium era capaz de provocar la muerte en los ratones infectados. Además, podía transmitirse de un roedor a otro en la misma jaula y aparentemente no representaba peligro para los humanos ni para el ganado. En 1892, los campos de Tesalia estaban siendo devastados por plagas de ratones y, sin encontrar una solución, recurrieron a científicos prominentes de la época en busca de respuestas. Pasteur compartió con el gobierno griego el trabajo de Loeffler y, sin demora, este último viajó a los campos para utilizar a S. Typhimurium como agente de control de plagas. Trozos de pan se empaparon en agua contaminada con la bacteria y se dispersaron por los campos, lo que resultó en una considerable cantidad de ratones muertos. Esto marcó el éxito tanto para Loeffler como para los griegos.
Después de este logro, otros microorganismos comenzaron a utilizarse para el control de plagas; sin embargo, la gradual comprensión de que Salmonella sp. está lejos de ser inofensiva para los humanos, ha llevado a su desuso.
Construyendo la armonía de las ilusiones
S. Typhimurium fue relegada al anecdotario de las armas olvidadas en la lucha contra las plagas; sin embargo, mientras estos ideales utilitarios se desdibujaban, un patógeno emergía. En la década de los 20s comenzó a manifestarse como un problema de salud en los seres humanos, causando infecciones gastrointestinales. Existían múltiples cepas aisladas de esta bacteria, no obstante, estudiarlas todas resultaba difícil, lo que llevó a los investigadores a centrarse en unas pocas a las que consideraron cepas prototipo o de referencia, un enfoque conocido como reduccionismo.
Aunque esta estrategia facilitó el trabajo, y ayudó a ir acumulando conocimiento que nos ha permitido ir comprendiendo, cada vez con mayor profundidad, la genética bacteriana y los mecanismos de infección de S. Typhimurium, perdió de vista el hecho de que las bacterias son seres muy dinámicos que poseen un genoma altamente plástico, capaces de evolucionar y adaptarse a diferentes entornos, por lo que una cepa no siempre sería idéntica a otra. Así, durante muchos años, solo se estudiaron las cepas que los grandes grupos de investigación consideraron relevantes para el avance del campo. Esto creo una realidad alterna, una armonía de las ilusiones que aún muchos investigadores comparten.
Rompiendo la armonía de las ilusiones
Los colectivos de pensamiento limitaron la posibilidad de que investigadores con diferentes perspectivas influyeran en el estudio de S. Typhimurium. Sin embargo, la era de la genómica y la secuenciación masiva del ADN de microorganismos ha comenzado a rasgar esta armonía de las ilusiones, revelando a S. Typhimurium más diversa y continuamente cambiante de lo que jamás imaginamos.
Técnicas como el Multi locus Sequence Typing (MLST), se han usado para identificar y clasificar las variantes de S. Typhimurium. El genoma de una bacteria es como una partitura, un conjunto de notas que conforman su identidad y le dan una melodía; en lugar de notas, estos genes están compuestos de secuencias genéticas específicas que varían entre bacterias. Así, el MLST armoniza las “notas” genéticas de diferentes cepas bacterianas, permitiéndonos comparar sus secuencias e identificar variaciones especificas en las “notas”, que le dan a cada grupo de cepas melodías únicas a las cuales se les denomina genotipos (ST).
Los primeros hallazgos revelaron que el genotipo más abundante en el mundo es el ST19. Este genotipo engloba a todas las cepas prototipo estudiadas en las últimas ocho décadas. Con el tiempo, otros genotipos han cobrado importancia, ya sea por su repentina abundancia, su resistencia a los antimicrobianos o su habilidad para causar enfermedades graves. Ejemplos notables incluyen los genotipos ST34 y ST313.
Estos genotipos contienen variaciones genéticas más allá del MLST y se han posicionado como riesgos a la salud pública. El genotipo ST34 es uno de los principales agentes causales de gastroenteritis e infecciones invasivas a nivel mundial y ha sido responsable de múltiples brotes en la última década, convirtiéndose en una cepa pandémica. Por otro lado, el ST313 se encuentra principalmente en África y algunas de sus cepas están asociadas con infecciones invasivas severas en personas con VIH, malaria o desnutrición. Se considera que este genotipo ha evolucionado hacia un estilo de vida antropogénico, mostrando gran capacidad de adaptación.
No obstante, los genotipos o STs de interés no se agotan ahí. En nuestra propia travesía, un genotipo específico llamó nuestra atención. Durante una vigilancia epidemiológica realizada en México en los albores del siglo XXI, se aislaron cepas de S. Typhimurium resistentes a varios antibióticos que, por coincidencias fortuitas y estrechas redes de colaboración, llegaron a manos de Edmundo Calva.
Fue Edmundo, quien impulsado por su curiosidad e instinto científico y un diálogo con un colega acerca de un nuevo método de subclasificación de cepas, propuso analizarlas usando el MLST. La sorpresa fue mayúscula al descubrir que la mayoría de las cepas pertenecían a un ST antes no descrito, el 213, y más aún porque algunas de estas cepas fueron aisladas de personas enfermas a causa de infecciones invasivas severas, incluso en individuos previamente sanos que no estaban inmunocomprometidos (Figura 3).
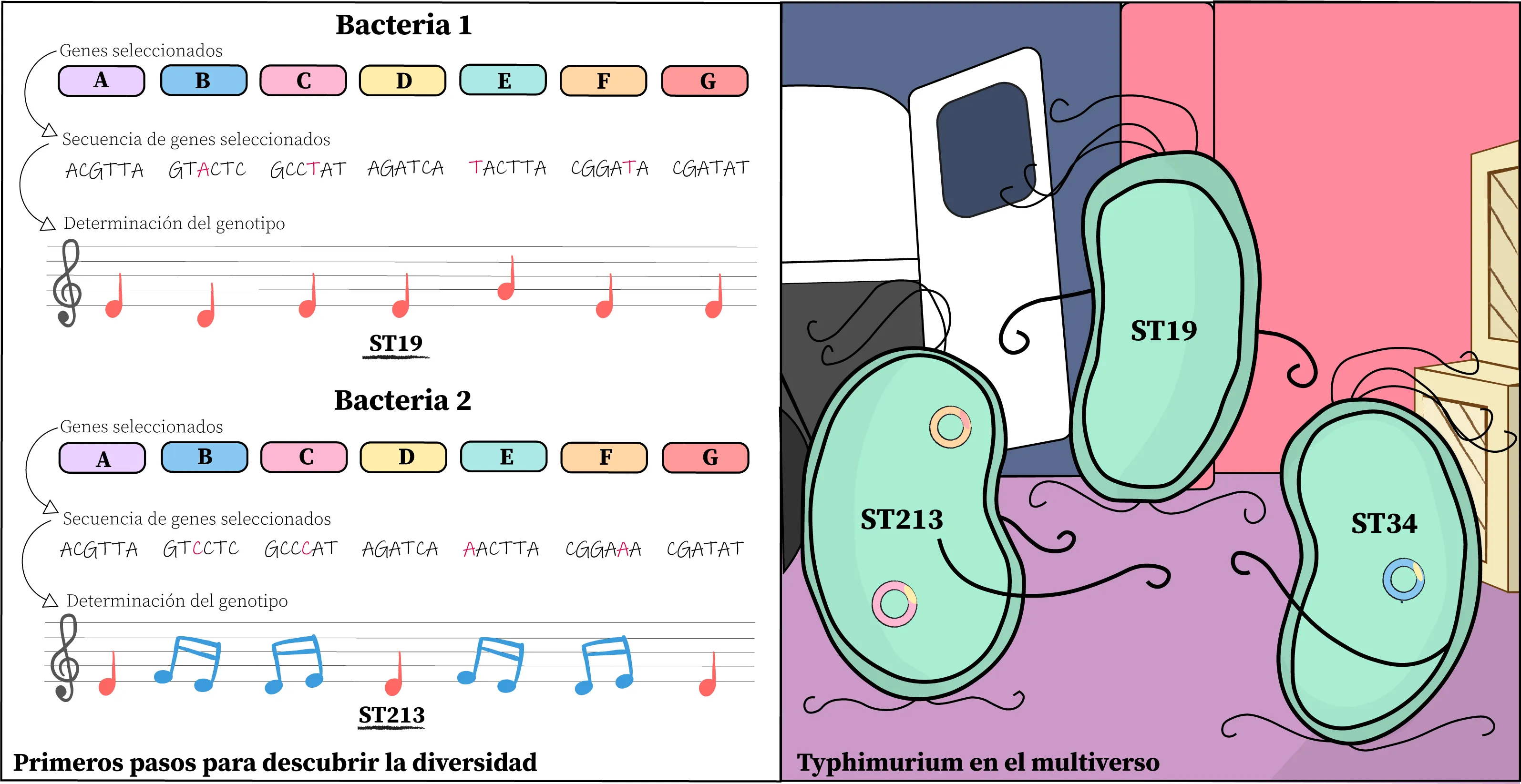
Figura 3. A través de la técnica del MLST, los científicos pueden clasificar a las bacterias en genotipos, y así cómo los músicos pueden identificar diferentes estilos y géneros musicales según las variaciones de las partituras, esta técnica permite un primer acercamiento a la exploración de la diversidad genética entre las bacterias, y ayuda a rastrear su evolución y diseminación.
Así comenzó esta historia que, durante casi 15 años, nos ha llevado a intentar revelar los misterios del genotipo ST213 de S. Typhimurium. Hoy en día, sabemos que este genotipo es tan singular como sus contrapartes emergentes antes mencionadas, con un genoma accesorio característico, y resistencia a múltiples antibióticos.
Hemos descubierto que las cepas ST213 aisladas en Norteamérica son distintas a las que se encuentran en Europa, un recordatorio tangible de la diversidad que esculpe la historia de S. Typhimurium. Además, algunas cepas, aunque pertenecen al mismo ST y fueron aisladas en la misma región geográfica, presentan comportamientos diferentes durante la formación de biopelículas, lo cual evidencia diversidad fenotípica intra-genotipo.
Pero eso no es todo, a pesar de estar vinculadas con infecciones graves, estas cepas se internalizan en menor medida en células humanas cultivadas en el laboratorio, a diferencia de las cepas prototipo ampliamente estudiadas; no obstante, son capaces de alcanzar altas tasas de replicación dentro de estas células. Este escenario abre ventanas a la especulación: ¿Será ésta otra estrategia de S. Typhimurium para pasar desapercibida en el cuerpo humano y poder causar infecciones más prolongadas?, si éste es el caso, los mecanismos que llevan a las cepas ST213 a actuar de esta manera, tan distinta de las cepas prototipo, están aún por descubrirse.
Mientras continuamos descifrando la compleja historia de estas cepas, es esencial mantener una vigilancia constante, ya que podrían extender su distribución, haciendo más difícil su control. Además, es importante continuar con la investigación rigurosa para avanzar hacia un entendimiento más profundo de su impacto potencial en la salud humana. Y, así, en el futuro desarrollar estrategias efectivas para el diagnóstico, prevención y tratamiento de las infecciones asociadas a este genotipo.
Corolario
Los pasajes inexplorados nos desafían a seguir investigando, a continuar dando voz a la historia evolutiva de S. Typhimurium ST213, recordando siempre que este conocimiento se ha logrado a hombros de gigantes y que los avances en la investigación actual serían imposibles sin los científicos que nos precedieron.
Sin lugar a duda, la historia de las S. Typhimurium ST213 no habría sido posible sin Edmundo Calva, un apasionado impulsor de la ciencia revolucionaria. Su legado y visión nos han dejado una enseñanza invaluable: explorar los senderos más inciertos para iluminar lo que antes estaba sumido en la oscuridad. En su memoria, buscaremos desafiar los límites de la armonía de las ilusiones para construir un sistema diferente de pensamiento en la nueva era de la bacteriología y la genómica.
Lecturas sugeridas
- Soto González, S. (2022). Tratamientos prometedores contra las bacterias resistentes: investigación de nuevos antibióticos y alternativas para las infecciones microbianas. Blog ISGlobal. https://www.isglobal.org/ca/healthisglobal/-/custom-blog-portlet/tratamientos-prometedores-contra-las-bacterias-resistentes-investigacion-de-nuevos-antibioticos-y-alternativas-para-las-infecciones-microbianas/90586/0
- Bonfil Olivera, M. (2004). ¿Somos bacterias?, ¿Cómo ves? (No. 67). Ojo de mosca No.67 - Revista ¿Cómo ves? - Dirección General de Divulgación de la Ciencia de la UNAM
- Calva, E. (2001). El genoma humano. ¿Cómo ves? (No. 37). https://www.studocu.com/es-mx/document/universidad-nacional-autonoma-de-mexico/bioquimica/el-genoma-humano-apuntes-genetica/6028684
- Fleck, L. (1935). La génesis y el desarrollo de un hecho científico. Alianza Editorial.
Comparte este artículo en redes sociales

Acerca de los autores
La M. en C. Isela Serrano Fujarte cursó sus estudios de Maestría en Ciencias (Biología) en la Universidad de Guanajuato y actualmente se encuentra cursando el Doctorado en Ciencias Bioquímicas en el Instituto de Biotecnología, UNAM. Su trabajo explora la diversidad de las cepas de S. Typhimurium ST213. La Biól. Jimena Berenice García Domínguez es egresada de la Facultad de Ciencias de la UNAM, realizó su tesis de Licenciatura estudiando las cepas de S. Typhimurium ST213. Actualmente cursa la Maestría en Ciencias Bioquímicas en el Instituto de Investigaciones Biomédicas, UNAM. El Dr. José Luis Puente García es líder académico en el Departamento de Microbiología Molecular del Instituto de Biotecnología, UNAM. Su grupo de investigación estudia los mecanismos moleculares que regulan la expresión de genes de virulencia en bacterias enteropatógenas.
Contacto: isela.serrano@ibt.unam.mx; jimena.garcia@ibt.unam.mx; jose.puente@ibt.unam.mx