Los modernos cazadores de microbios
Maricela Carrera Reyna y Rosa María Gutiérrez Ríos
En el siglo XVII se cultivaron los primeros microorganismos. Sin embargo, muchos de ellos viven en condiciones que aún no podemos recrear en el laboratorio, ¡Son huéspedes muy exigentes! Esta dificultad impulsó el desarrollo de métodos moleculares y computacionales para su estudio y exploración.
¿Cómo sabemos que existen microorganismos que no pueden cultivarse en condiciones artificiales en el laboratorio?
El primer indicio de la existencia de microorganismos no cultivables surgió cuando los científicos Staley y Konopka, en 1985, observaron una discrepancia entre el número de microorganismos provenientes de entornos naturales observados bajo el microscopio y el número de colonias que se obtenían al sembrar la muestra ambiental en medios de cultivo de laboratorio. Este fenómeno se denominó la “anomalía del recuento de grandes placas” (Figura 1).
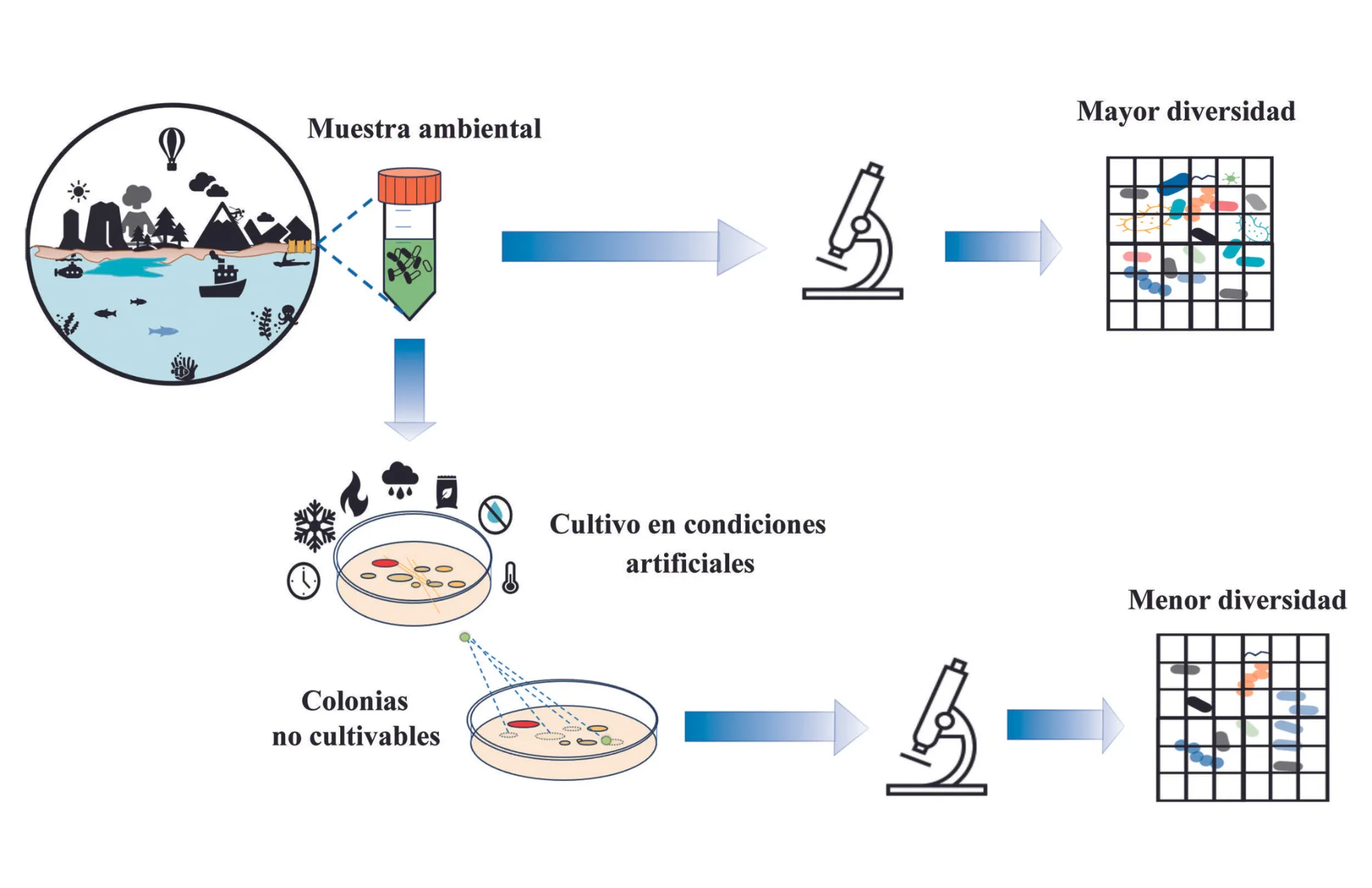
Figura 1. Anomalía de las grandes placas. Diferencias en el número de organismos observados al microscopio derivados de una muestra ambiental y un crecimiento microbiano en caja de Petri con agar y un medio nutritivo, en condiciones artificiales de laboratorio del mismo origen.
Una de las razones de esta discrepancia se asocia a las condiciones ambientales en las que habitan, como la temperatura, acidez del medio, y combinaciones particulares de nutrientes o requerimientos de oxígeno. En algunos casos, los microorganismos crecen lentamente en condiciones de laboratorio, por lo que no se pueden observar en cultivos a corto plazo (horas o días). Además de que identificarlos por su aspecto físico, es decir el fenotipo , es también un reto, ya que son indistinguibles a nuestro ojos.
Las relaciones que los microorganismos establecen para sobrevivir en su ambiente pueden compararse con relaciones sociales, donde cada acción tiene repercusiones en la comunidad. Las acciones que ejercen los microorganismos sobre su comunidad son señales químicas que estos interpretan como estímulos positivos para el crecimiento o como agresiones, que pueden llegar a ser letales para algunos miembros de la comunidad. Por ejemplo, algunas bacterias producen antibióticos naturales o moléculas proteicas llamadas bacteriocinas, que son estímulos agresores, capaces de inhibir el crecimiento de otros miembros de la comunidad.
Por otro lado, las bacterias pueden crear superficies compuestas por azúcares conocidos como exopolisacáridos en donde las comunidades microbianas se adhieren y multiplican creando una estructura llamada biopelícula (Figura 2). Los microorganismos creciendo en las biopelículas se comunican a través de señales químicas que también regulan la estructura de la comunidad microbiana. La biopelícula es una herramienta que permite a los microorganismos colonizar superficies como el agua, suelo, y materiales quirúrgicos.
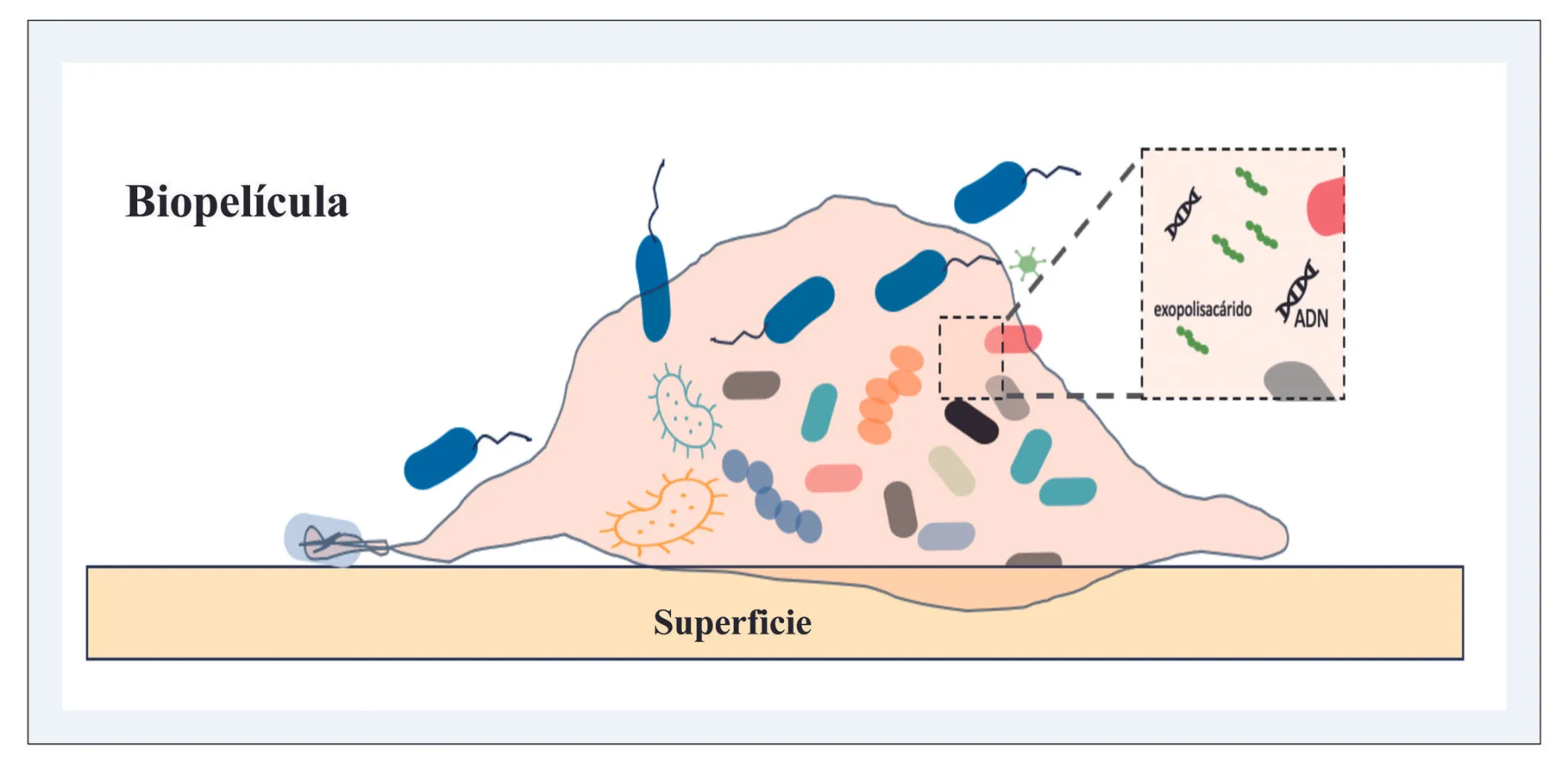
Figura 2. Biopelículas. Estructura de una comunidad bacteriana habitando una matriz de exopolisacáridos.
Como nuestros lectores podrán notar, muchas condiciones y señales no pueden replicarse fácilmente en el laboratorio. Y a pesar de comprender algunos de los mecanismos de interacción microbiana en su entorno natural, todavía existe un vasto terreno de conocimiento por explorar que impide actualmente cultivar cualquier microorganismo en laboratorio. Entonces, ¿cómo podemos reconocer y estudiar experimentalmente estos microorganismos?
A la caza de microbios no cultivables
Los conocimientos desarrollados entre 1930 y 1950 en la biología molecular y la bioquímica, así como el desarrollo de las ciencias físicas y la computación en años posteriores, nos permitieron explicar cómo se codifica, organiza, transmite y evoluciona la información genética. Por lo que, no era descabellado suponer qué piezas de esta información podrían ayudarnos a estudiar a los organismos no cultivables.
En 1970, Carl Woese y George E. Fox analizaron segmentos de un pequeño trozo de ADN codificando al gen 16S ribosomal, presente en la subunidad pequeña del ribosoma celular, que es la molécula encargada de traducir los mensajes codificados en el ADN en proteínas. El análisis de este gen reveló variaciones suficientes para establecer una clasificación que permite distinguir entre tipos de bacterias e incluso dio lugar al descubrimiento de un nuevo grupo: las arqueobacterias.
Actualmente, sin necesidad de cultivar en el laboratorio, podemos determinar qué microorganismos habitan en el ambiente gracias al desarrollo de la técnica conocida como reacción en cadena de la polimerasa (PCR por sus siglas en inglés). Esta innovación ha sido la base para la amplificación de segmentos de ADN como molde, posibilitando la obtención masiva del material genético de microorganismos presentes en muestras ambientales, tales como los océanos, la microbiota de distintos organismos, el suelo y el aire. Ahora, es factible conocer la diversidad microbiana de tales muestras sin recurrir al cultivo artificial en laboratorio. Las técnicas basadas en la secuenciación masiva permiten leer el ADN de muchos microorganismos a la vez y clasificarlos, lo que da a los científicos una imagen mucho más completa de la diversidad microbiana en un entorno (Figura 3).
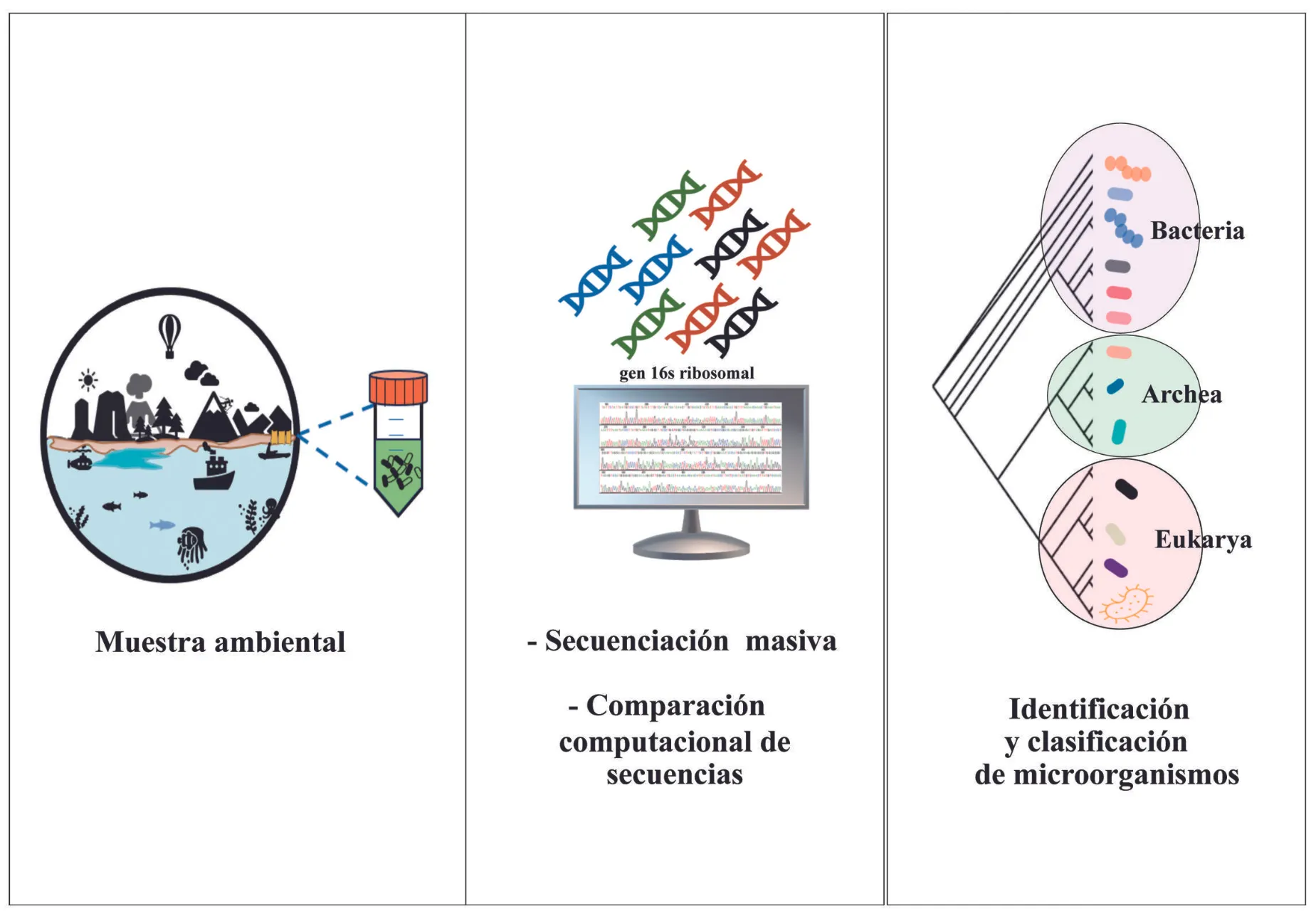
Figura 3. Del ambiente a la clasificación de los microorganismos que lo habitan.
Explorando los ecosistemas
Como ya se describió, el uso del gen 16S ribosomal ha permitido identificar un gran número de bacterias y arqueas no cultivables en diversos ecosistemas. Por su parte, el gen 18S, que es un componente de la subunidad pequeña de los ribosomas de organismos eucariontes, ha sido útil para identificar organismos fúngicos microscópicos ocultos en muestras ambientales. Gracias a estas nuevas técnicas, los científicos han descubierto un mundo completamente nuevo de microorganismos que antes eran invisibles.
La caza de microorganismos no cultivables identificados con las tecnologías de secuenciación masiva nos ha ayudado a comprender el papel que juegan en muchos ecosistemas. Un ejemplo de esto, lo vimos en una de las catástrofes ambientales más grandes de la historia: el derrame de petrolero ocurrido en el golfo de México en la plataforma Deep Water Horizon el 22 de abril de 2010 y que se estima vertió 779 mil toneladas de petróleo crudo al mar. La secuenciación del material genético extraído de muestras del sitio contaminado reveló la presencia de genes en los genomas de microorganismos del Golfo de México, codificando proteínas con la capacidad de degradar hidrocarburos del petróleo (Figura 4). La identificación de estos genes, junto con mediciones que indicaron el fraccionamiento de los hidrocarburos, demostró que, aunque no se puedan cultivar a todos los microorganismos, el golfo de México alberga organismos nativos que han desarrollado la capacidad de degradar estos compuestos tras estar en constante contacto con ellos. Lamentablemente, a pesar de la ayuda de nuestros amigos invisibles, llevará mucho tiempo restaurar en su totalidad a este gran ecosistema del golfo de México.
En el Instituto de Biotecnología (IBt), se trabaja buscando aliados bacterianos no solo para enfrentar desastres derivados de derrames de petróleo (consultar ATLAS de bacterias del golfo de México), sino también otros problemas ambientales. Estos organismos ayudan a superar adversidades que afectan a los ecosistemas y a los organismos que los habitan. Así, las investigaciones de distintos grupos en el IBt buscan aprovechar el potencial de los microorganismos para mitigar diversos tipos de desastres ambientales y curar enfermedades subrayando la importancia de nuestros invisibles y poderosos aliados en la lucha por un mundo más sostenible.
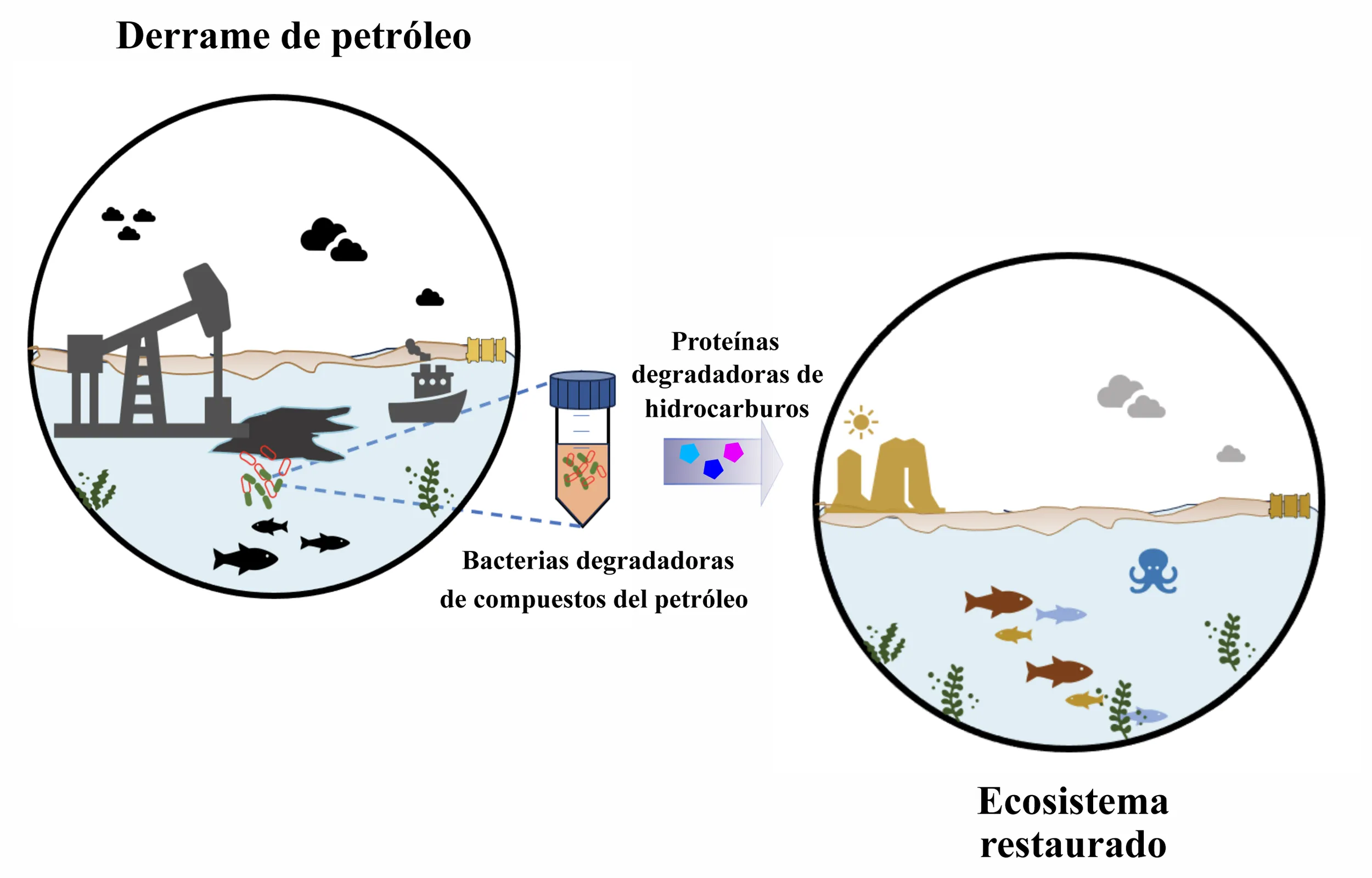
Figura 4. Microorganismos en acción. Limpiando un ambiente perturbado por un derrame de petróleo.
Lecturas recomendadas
- Kimes NE, Callaghan AV, Suflita JM, Morris PJ. Microbial transformation of the Deepwater Horizon oil spill-past, present, and future perspectives. Front Microbiol. 2014 Nov 18;5:603. PMID: 25477866; PMCID: PMC4235408. Resumen: En este artículo encontrarás información sobre cómo los nuevos cazadores de microbios encontraron evidencia de que con ayuda de los microorganismos y algunos métodos químicos los compuestos del petróleo con una composición química menos compleja habían sido degradados en el sitio del derrame en la plataforma Deepwater Horizon. También puntualiza que es posible que compuestos más recalcitrantes no puedan ser degradados por microorganismos. DOI: 10.3389/fmicb.2014.00603.
- Gutierrez-Rios, R.M., Juarez-Lopez,K., Loza-Roman,J.A., Merino-Perez,E., Pardo-Lopez,L., Sanchez-Flores,F.A., Segovia-Forcella,L.P., CICESE, CINVESTAV (2021). Atlas de línea base ambiental del Golfo de México.Tomo X. Distribución de bacterias. atlascigom.cicese.mx/es/dataset/libro-atlas-tomo-10. Resumen: En este libro encontrarás los resultados de los esfuerzos hechos por científicos mexicanos de tres instituciones nacionales, incluidos investigadores del Instituto de Biotecnología de la UNAM, para caracterizar a las bacterias del golfo de México con y sin capacidades degradadoras de hidrocarburos del petróleo.
- Harwani D. The Great Plate Count Anomaly and the Unculturable Bacteria. IJSR - International Journal of Scientific Research. 2013 Sep 2(9) ISSN No 2277 – 8179. Resumen: En esta publicación se muestran evidencias de que muchas células bacterianas son metabólicamente activas, aunque no puedan crecer en medios artificiales en el laboratorio.
- Balch WE, Magrum LJ, Fox GE, Wolfe RS, Woese CR. An ancient divergence among the bacteria. J Mol Evol. 1977 Aug 5;9(4):305-11. PMID: 408502. Resumen: En esta publicación se discute el uso del gene 16S ribosomal como una macromolécula común a todas las células, que por sus características es utilizada para la identificación de especies microbianas y su clasificación en el árbol de la vida. En este artículo también demuestran como dos especies de microorganismos capaces de producir metano a partir de dióxido de carbono presentan cambios en la secuencia del gene 16S ribosomal que al clasificarlas las separa de las bacterias. DOI: 10.1007/BF01796092.
- “Por fortuna existen los microbios”, de J. Caballero acmor.org/articulos-anteriores/por-fortuna-existen-los-microbios.
- Paul de Kruif. Los cazadores de microbios. Editora Géminis.1997. Primera edición. ISBN 978-9962-806-45-5
Comparte este artículo en redes sociales

Acerca de los autores
Maricela Carrera Reyna: Estudiante de doctorado con interés en la aplicación de métodos computacionales para la exploración de genomas microbianos. Entusiasta de la enseñanza de distintos lenguajes de programación y socia fundadora de PyLadies Cuernavaca. La Dra. Rosa María Gutiérrez Ríos es investigadora en el área de genómica y biología computacional. Sus investigaciones están enfocadas en entender a nivel de sistema, las interacciones genético-metabólicas que modulan la expresión de los genes en procariontes cultivables y no cultivables. Autora de correspondencia.
Contacto: maricela.carrera@ibt.unam.mx, rosa.gutierrez@ibt.unam.mx

