Un laboratorio navegante para descifrar los misterios de bacterias marinas
Laura Cristina Martínez Martínez y Karla Sofía Millán López
El mar es un misterio en donde aún hay cosas por descubrir, pero estudiar las profundidades puede ser algo complicado. En el Golfo de México existe un área con una profundidad entre 3 mil 750 y 4 mil 384 metros con un promedio de 1,615 metros, conocida como la fosa de Sigsbee. Este ambiente tan especial ha acaparado la atención del grupo de la Dra. Elva Escobar Briones, quien pertenece al Instituto de Ciencias del Mar y Limnología de la UNAM. De manera recurrente, a bordo del buque oceanográfico Justo Sierra, la Dra. Escobar junto con una tripulación de diferentes científicos realiza campañas para estudiar la fosa de Sigsbee. Cuando el buque se posiciona en la zona de interés, se toman muestras de agua y sedimentos empleando diferentes equipos especializados.
Este año, el grupo de científicos del Laboratorio de Biotecnología Marina del Instituto de Biotecnología (IBt) de la UNAM, dirigidos por la Dra. Liliana Pardo López, tuvo la oportunidad de participar en la campaña oceanográfica “SIGSBEE-2024", realizada del 11 al 15 de junio del 2024. En esta campaña, los científicos del grupo de la Dra. Pardo se dieron a la tarea de conocer cuáles son las bacterias que habitan este ambiente marino, por lo que en este artículo se relata la forma en la que se logró esta hazaña.
¿Alguna vez te has preguntado si en el mar habitan organismos que no vemos? La respuesta es sí, existen organismos microscópicos que nuestros ojos no alcanzan a ver, como lo son las bacterias. Recrear las condiciones para estudiar bacterias marinas es algo complejo, pues implica la puesta en marcha de condiciones especiales en el laboratorio, por ejemplo, de temperatura, salinidad, disponibilidad del oxígeno y presión, por lo que muchas bacterias no llegan vivas al laboratorio y no pueden identificarse, convirtiéndolas en un misterio.
En México, el Laboratorio de Biotecnología Marina del IBt tiene como objetivo resolver estos misterios y en los últimos años, se han enfocado en describir las comunidades microbianas de la zona sur del Golfo de México y descubrir las características que las hacen especiales para sobrevivir en las profundidades, mediante cruceros oceanográficos en donde se analizan muestras de agua y sedimento marino. Entre sus hallazgos están la identificación de bacterias que pueden comer hidrocarburos, como el petróleo, y degradar diferentes tipos de plásticos, así como descifrar las posibles formas en las que lo logran [1]. Estas investigaciones las realizan utilizando estrategias moleculares en las cuales el ácido desoxirribonucleico, mejor conocido como ADN, es el protagonista.
Antes de zarpar
¿Imaginas un laboratorio en medio del mar? El buque Justo Sierra es un laboratorio andante donde se pueden realizar investigaciones y al mismo tiempo observar los increíbles amaneceres antes de comenzar el día o los atardeceres al terminarlo (Figura 1).

Figura 1. Amaneceres en el Justo Sierra. No se puede estar en un barco y no apreciar estos maravillosos paisajes.
En el caso de un laboratorio navegante, la planeación requiere semanas o meses de anticipación. Por esta razón, durante las semanas previas al comienzo del viaje, se guarda el material que se llevará a bordo, siempre asegurando que no falte nada mediante una revisión detallada a la bitácora del viaje, pues una vez en medio del mar es prácticamente imposible conseguir cualquier cosa. Antes de zarpar se acondiciona el laboratorio, distribuyendo las estaciones de trabajo de acuerdo con los experimentos planeados y asegurando con cuerdas los equipos a las mesas o a las paredes para evitar algún accidente debido al movimiento de las olas (Figura 2).

Figura 2. Adaptación del laboratorio de biotecnología a bordo del buque oceanográfico Justo Sierra.
En el buque oceanográfico existe una organización en donde toda la tripulación es asignada a una guardia específica. Los horarios son de 4 a 8, 8 a 12 y 12 a 4, tanto en la noche como en el día. Esto significa que se deben cumplir labores en la cubierta, en el laboratorio de registro central y cómputo y además el laboratorio de biología, espacio asignado especialmente para el laboratorio de biotecnología marina. Como puede verse, la organización es un punto vital para que todo salga bien.
Colecta de las muestras
Para estudiar las bacterias marinas, primero es importante tomar las muestras de sedimento y agua a diferentes profundidades con las que se va a trabajar. Para lograrlo, se usan equipos especiales. La obtención del sedimento marino se logra empleando un equipo llamado “multinucleador” el cual está conformado por 12 tubos de policarbonato que se entierran en el fondo marino, lo que permite capturar el sedimento y transportarlo a la cubierta en donde es seleccionado, procesado y almacenado; mientras que, para recuperar el agua se utiliza un equipo conocido como “Rosetta”, (para más detalles leer el artículo en este mismo número: “Buque oceanográfico Justo Sierra: Ciencia flotante en el golfo de México”).

Figura 3. Científicos trabajando en la colecta de las muestras. A la izquierda se puede observar el equipo empleado para recolectar el agua de mar (“Rosetta”) y a la derecha la recuperación del sedimento del fondo marino.
Más de 100 litros del agua recolectada son filtrados (un proceso que puede tardar más de 6 horas) con una membrana de tamaño de poro diminuto, tan pequeño que las bacterias no pueden pasar y quedan atrapadas en el filtro.
De forma cotidiana, los científicos del laboratorio de Biotecnología Marina del IBt realizan técnicas de biología molecular, microbiología y bioinformática. Sin embargo, en el buque se requieren de otras habilidades, por ejemplo, mayor actividad física, adaptarse a los movimientos del barco, ajustarse a diferentes horarios de trabajo, entre otras. Es importante resaltar la importancia del trabajo en equipo, dentro y fuera del laboratorio. En este caso, la cooperación permitió que los miembros con experiencia pudieran orientar a los novatos en el desarrollo de todas las actividades de la campaña oceanográfica.
El ADN en altamar
En trabajos previos del grupo de Biotecnología Marina del IBt, las muestras fueron tomadas en altamar y procesadas en tierra después de varios días de almacenamiento. Uno de los mayores problemas detectados con esta estrategia fue que el ADN bacteriano obtenido presentaba baja cantidad y calidad (aún no está muy claro por qué sucede). Para intentar hacer más eficiente el proceso, se desarrolló una nueva estrategia molecular a bordo del buque durante esta campaña, en donde el ADN se procesaría en el sitio.
El ADN es una molécula que sirve como un manual de instrucciones escrito en una especie de código, donde se almacena la información sobre cómo funcionan y se desarrollan los organismos vivos. El lenguaje en el que está “escrito” este manual implica la utilización de cuatro “caracteres”, conocidos como nucleótidos: Adenina (A); Timina (T); Guanina (G) y Citosina (C). Conocer el orden de los nucleótidos en las moléculas de ADN es de mucha importancia para descifrar estas instrucciones.
Lograr conocer el orden de los nucleótidos a lo largo de las moléculas de ADN, implica utilizar una técnica conocida como secuenciación de ADN, que busca determinar el orden preciso de los nucleótidos a través de técnicas moleculares muy especializada (para más detalles leer el artículo “Secuenciación Masiva de ADN” por el Dr. Ricardo Grande).
Gracias a esta herramienta se pueden identificar bacterias y organismos sin necesidad de cultivarlos en el laboratorio, lo cual se conoce como metagenómica. Esta técnica estudia el material genético total extraído directamente de una muestra. Es utilizada para estudiar comunidades específicas de microorganismos, como los que habitan en el suelo, en el fondo marino o en la piel humana. Para lograrlo, se apoya del uso de técnicas de biología molecular y bioinformática. Una ventaja es que se puede usar ADN de muestras tomadas directamente del ambiente, sin la necesidad de pasar por los medios de cultivos, permitiendo conocer casi todos los microorganismos presentes en el ambiente (para conocer más detalles, consultar el artículo “Los modernos cazadores de microbios”).
Recuperando el tesoro: obtención del ADN.
La campaña oceanográfica duró 5 días, de los cuales 2 se utilizaron para la navegación hasta la fosa de Sigsbee, en donde comenzó la recolección y procesamiento de muestras. Era crucial que los científicos a bordo actuaran con rapidez para garantizar que el ADN estuviera secuenciado para el momento en que se llegara a tierra.
Descifrar la identidad de las bacterias implica el uso de métodos de biología molecular, en el que se usan paquetes comerciales, para que la extracción del material genético sea lo más rápido y fácil posible. Esto implicó el trabajo de los cinco investigadores, quienes se pusieron manos a la obra, no sin antes tomar un par de horas para ir a la biblioteca y planear la estrategia para comenzar con la extracción.
Alrededor de las 4 pm empezaron con el proceso de extracción y para las 7 pm aseguraron la presencia de ADN usando una técnica conocida como electroforesis (Figura 4). Dado que aún faltaba preparar la molécula para la secuenciación, se establecieron turnos internos, para así poder balancear el trabajo y el descanso. Con esto, se lograron 24 horas continuas de trabajo efectivas. Los procesos en donde se requería menos movimiento se realizaban en una mesa estabilizadora, diseñada para contrarrestar el oleaje.
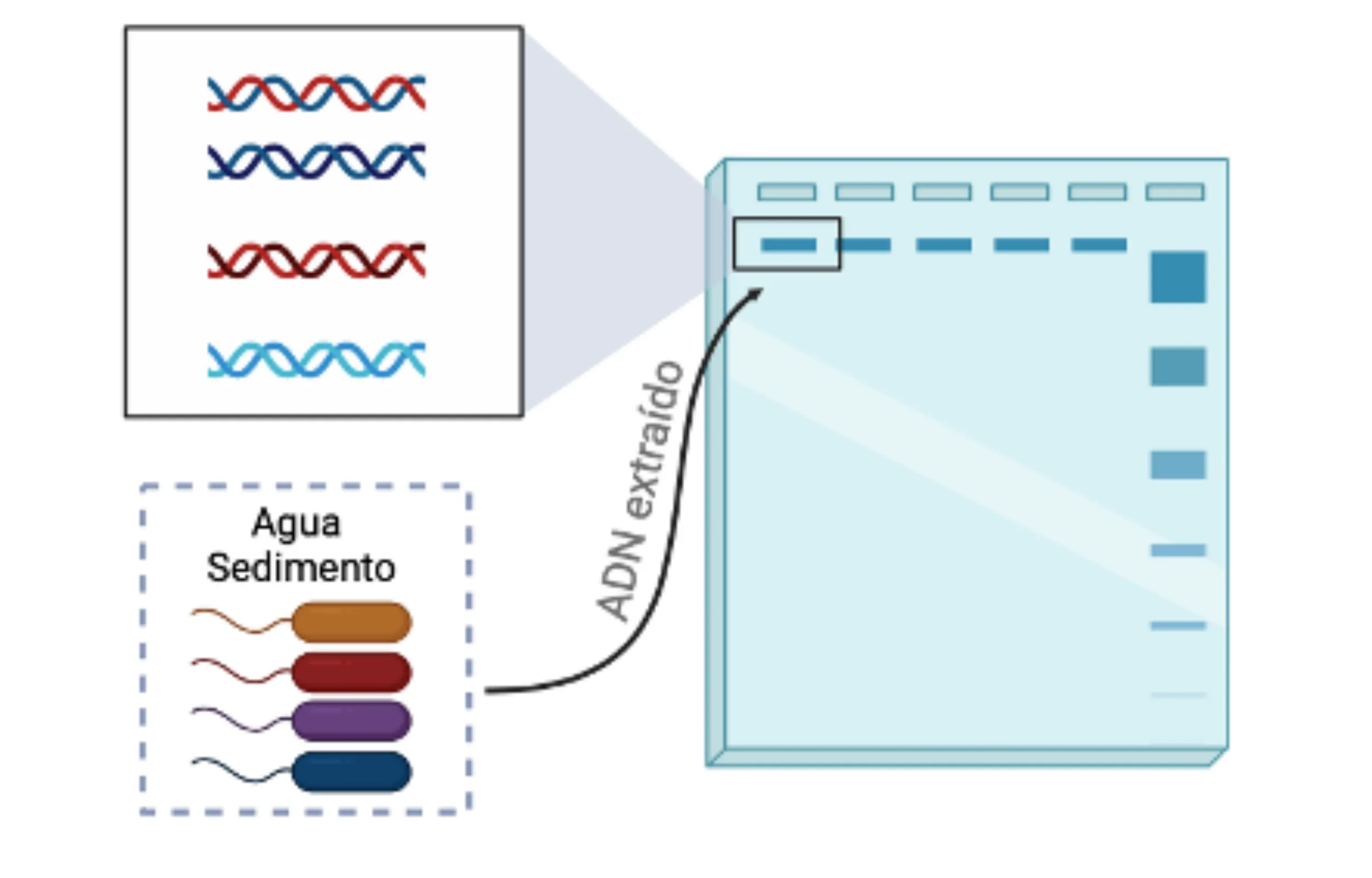
Figura 4.
Visualización de ADN. El material genético de cientos de bacterias que se obtiene después de la
extracción de las muestras ambientales fue visualizado utilizando un
Existen regiones del ADN denominadas marcadores moleculares, que pueden usarse como un tipo de código de barras. Similar a los que se usan al comprar en el supermercado. Con estos “códigos de barra”, se puede identificar a qué género pertenece la bacteria. Para el caso de la identificación y clasificación de las bacterias, el marcador más utilizado es un gen llamado “16S ribosomal”, que codifica para la subunidad pequeña del ribosoma y que, a lo largo de su secuencia, presenta cambios que se conservan entre miembros de un mismo género, pero que difieren cuando se compara contra otro género bacteriano. Estos “códigos de barra” no se pueden ver a simple vista, por lo que se usa la secuenciación para analizarlos.
En el barco se utilizó un secuenciador portátil llamado MinION, del tamaño de una tableta electrónica,
el cual trabaja con
La secuenciación de ADN a través de nanoporos implica la lectura del código de cadenas únicas de ADN a medida que se las
enlaza a través de poros extremadamente pequeños (nanoporos) insertados en una membrana. A medida que el ADN se desplaza
a través del poro, crea señales que se pueden convertir para leer cada base. Este enfoque ofrece un proceso rápido y de
bajo costo para estudiar largos tramos de ADN.

Figura 5. Secuenciador de ADN MinION. a) Las muestras son depositadas en el “ flow cell” el cual está dotado de cientos de poros disponibles para la secuenciación. b) Una vez iniciado el proceso puede ser visualizado en la pantalla del equipo en tiempo real, los cuadros en color verde en la imagen indican poros secuenciados adecuadamente. c) Manipulación de uno de los componentes del equipo minION en el barco.
El procesamiento del material genético para la secuenciación, así como el uso del MinION, fue algo novedoso para el grupo. Por esta razón, el Dr. Ricardo Grande, quien es técnico académico de la Unidad Universitaria de Secuenciación Masiva y Bioinformática les brindó una capacitación de todo el proceso en las semanas previas al viaje, siendo una parte crucial en esta travesía. En la figura 6 se muestra el proceso general utilizado para el procesamiento de las muestras en el barco.
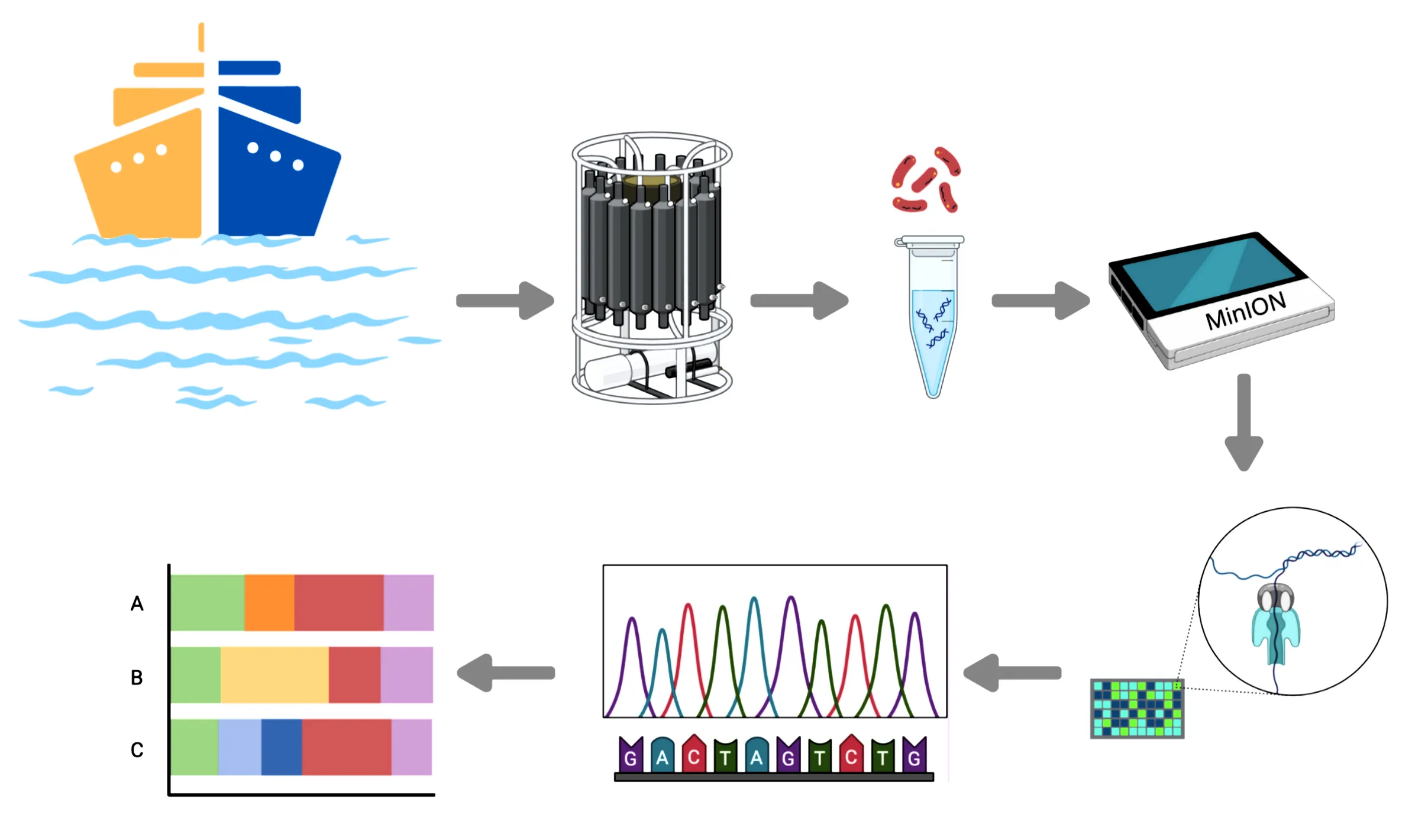
Figura 6. Diagrama general del proceso para conocer la diversidad bacteriana de la fosa de Sigsbee. Se colectan las muestras en altamar, se realiza la extracción de ADN y se procesa. Posteriormente se coloca en el secuenciador portátil, en donde el ADN pasa por unos poros diminutos que arrojará el orden de los nucleótidos de la muestra. Estos resultados son procesados computacionalmente para poder obtener la cantidad y la identidad de las bacterias que habitan la zona muestreada.
Descifrando el misterio
Esta aventura fue exitosa y empleando el MinION se logró secuenciar el ADN de bacterias de las muestras obtenidas a bordo del Justo Sierra.
El trabajo continúa una vez en tierra, en donde los datos de la secuenciación deben analizarse con programas bioinformáticos (los cuales requieren mucho poder computacional). La información descifrada se utilizará para saber cuáles son las bacterias que habitan el Golfo de México e identificar las más abundantes.
El establecimiento de esta estrategia de secuenciación en mares mexicanos permitirá que en el futuro se pueda identificar indirectamente si el mar está perturbado por algún contaminante, mediante diferentes muestreos a través del tiempo. Además, esta tecnología abre las puertas de su aplicación en diferentes ambientes.
La secuenciación de ADN en altamar ya se ha hecho en otras partes del mundo, pero en México no se había aplicado hasta el momento. Establecer un laboratorio que contemple la extracción y secuenciación de ADN en medio del mar fue un gran reto pues no se cuentan con los mismos equipos que en tierra, por lo que la planeación y el ingenio para resolver los problemas que se presenten es crucial. A pesar de los obstáculos, estos exploradores del mar, esperan continuar con estas travesías y mejorarlas con el tiempo. Solo así se podrá obtener más conocimientos sobre los microorganismos que habitan el gran Golfo de México.
Referencias
- Rodríguez-Salazar, J., Loza, A., Ornelas-Ocampo, K., Gutierrez-Rios, R. M., & Pardo-López, L. (2021). Bacteria From the Southern Gulf of Mexico: Baseline, Diversity, Hydrocarbon-Degrading Potential and Future Applications. Frontiers In Marine Science, 8. DOI: 10.3389/fmars.2021.625477
Lecturas recomendadas
- Adaya, L., Soto A., Ponce F. y Pardo L. (2024). Buque oceanográfico Justo Sierra: ciencia flotante en el golfo de México. Biotecnología en Movimiento. 1 (38). https://biotecmov.ibt.unam.mx/numeros/38/1.html
- Carrera R. y Gutiérrez R. (2024). Los modernos cazadores de microbios. Biotecnología en Movimiento. 4(37). https://biotecmov.ibt.unam.mx/numeros/37/4.html
- Grande, R. Secuenciación Masiva de ADN. (2018). Biotecnología en Movimiento. 4(13). https://biotecmov.ibt.unam.mx/services/pdfDownloader.php?id=MTMqKl8qKjQ=
Comparte este artículo en redes sociales

Acerca de las autoras
Karla Sofía Millán López es bióloga egresada de la Universidad Autónoma del Estado de Morelos, cursó la maestría en el IBt y actualmente realiza el doctorado enfocando sus estudios en la generación de consorcios bacterianos sintéticos para degradar hidrocarburos en arenas costeras en el Laboratorio de Biotecnología Marina del IBt-UNAM. Laura Cristina Martínez Martínez es Licenciada en Biotecnología Genómica por la Universidad Autónoma de Nuevo León. Está inscrita en el programa de Maestría en Ciencias Bioquímicas de la UNAM y su investigación se centra en evaluar la degradación de hidrocarburos de un consorcio bacteriano sintético en agua de mar en el Laboratorio de Biotecnología Marina del IBt-UNAM.
Contacto: cristina.martinez@ibt.unam.mx; karla.millan@ibt.unam.mx